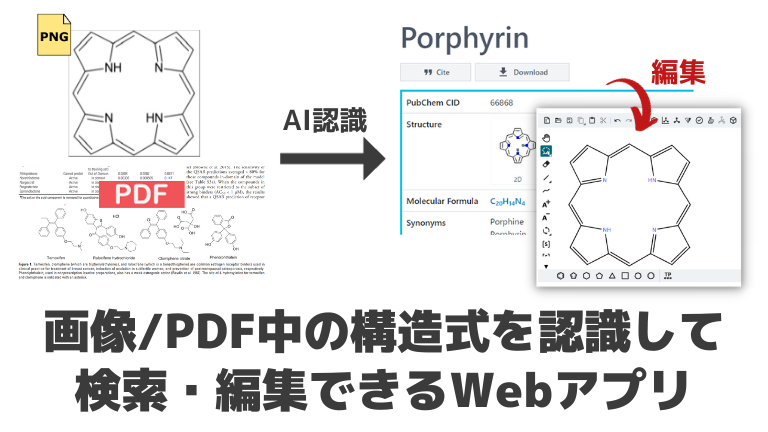
手元にある構造式からその物質の名前や詳細を調べたい時には、化合物検索サイトの構造式検索機能を使うのが一般的です。
リンク①:構造式を描いて物質を検索できるサイト
リンク②:描いた構造式と同一or近い物質のWikipedia記事を検索できるサイト
しかし調べたい構造式が何個もある場合や、描くのが面倒な大きい構造式の場合はエディタにいちいち描く作業が大変になります。
そのような場合は、画像中の構造式を認識して物質を検索できるサービス 「DECIMER web app」が大変便利です。
構造式をうまく認識しれくれれば面倒なエディタ入力作業が不要になるため高速で検索できます。
認識が多少誤っている場合もエディタから一部分だけ修正できるため、一から入力するよりは楽です。
特に凄いのが、PDFをアップするとPDF中の全ての構造式を自動で認識して一つ一つ検索用リンクを生成してくれることです。
知らない構造式から物質を調べる作業が爆速化できます。
しかも構造式の検索だけでなく、画像認識した構造式に対して以下の機能が利用できます。
- 認識した構造式を SMILES記法 に変換
- 認識した構造式を好きなファイル形式で保存 (.mol 形式 / .svg ベクター画像 / .png 画像…)
- 構造式から IUPAC名 に変換
- 構造式を検索用文字列に変換 (InChI)
- 構造式の認識結果が誤っていても、類似した構造式の化合物を探せる
- 認識した構造式をその場でエディタ編集し、手間をかけず構造式を描く
以下、DECIMER web app の基本的な使い方や各機能について説明します。
基本操作:画像から構造式を認識して検索
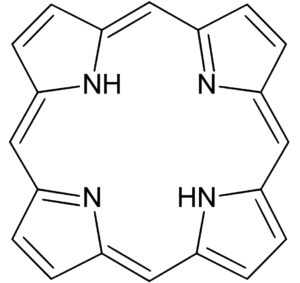
今回、こちらの構造式の画像で試してみます。(ポルフィリン)
まず、以下のリンクから DECIMER web app のサイトに移動します。
赤矢印で示したボタンのところに構造式が写った画像ファイルをドラッグ&ドロップするか、ボタンを押してファイル選択ダイアログを表示させてそこから画像を選んでください。
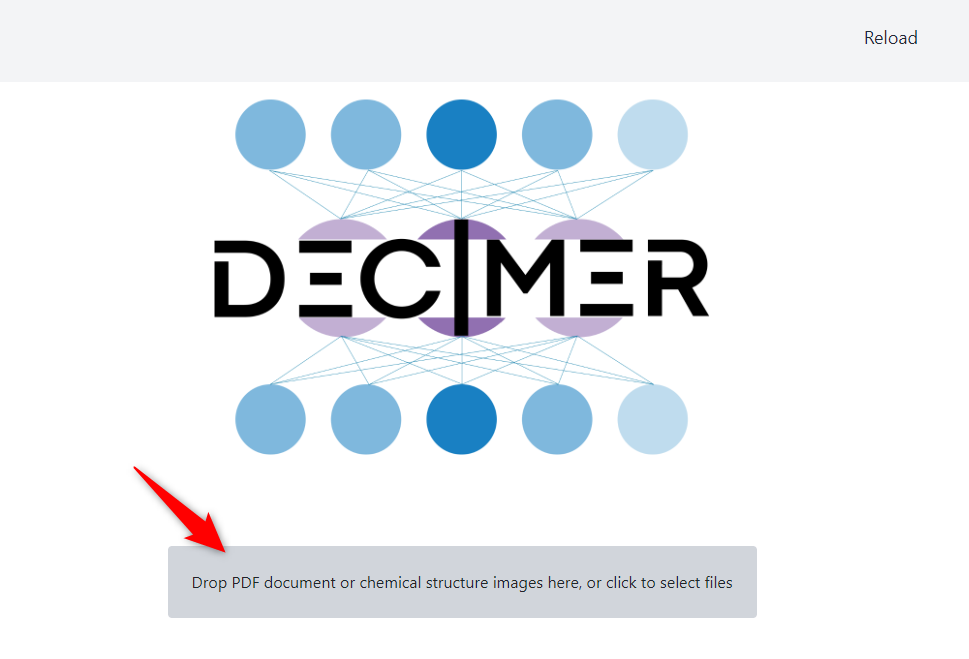
なお、構造式の画像は複数枚同時認識に対応しています。
まとめてアップすればそれぞれの画像について構造式認識が行われます。
画像アップロード後、認識処理が行われるのでしばらく待ちます。
処理が終わると認識結果が表示され、構造式検索を含めた色々な機能が利用できるようになります。
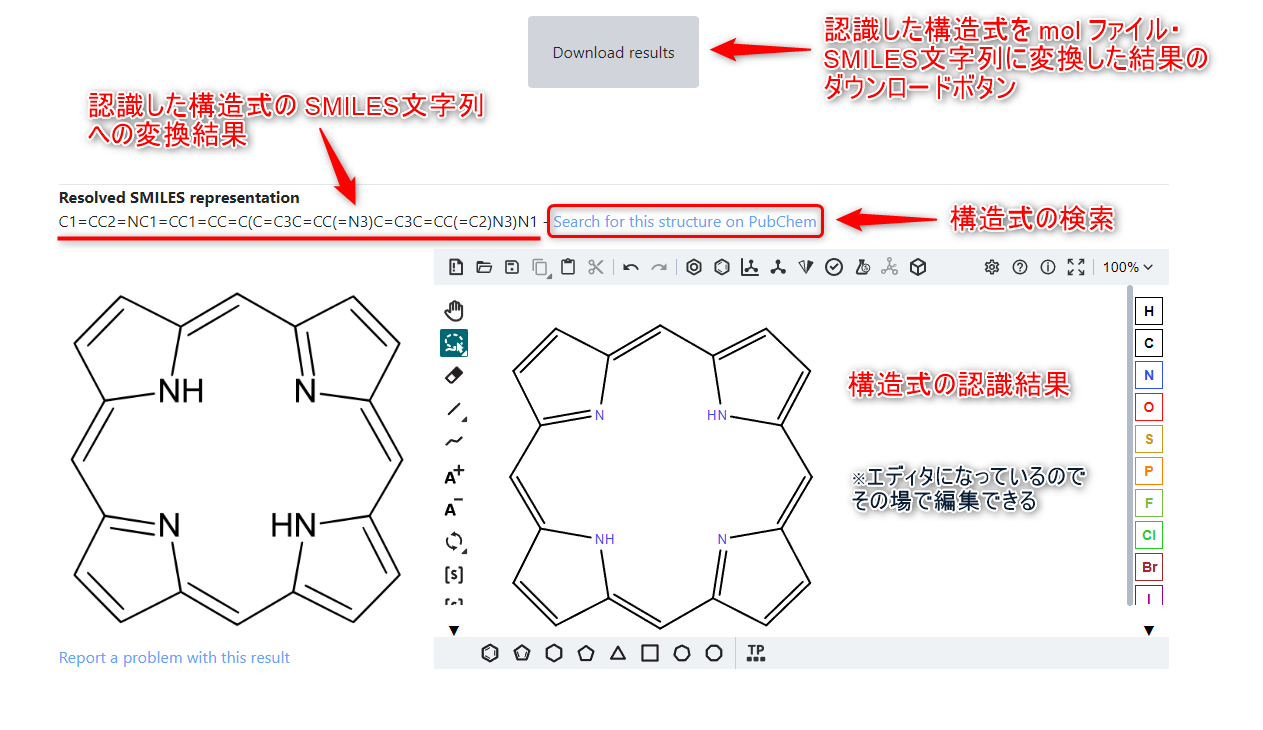
※上記画像のように、認識した構造式の向きは元の画像と異なる場合があります。(構造としては同一であり、正しく認識できています)
認識した構造式の検索
構造式エディタの上にある「Search for this structure on PubChem」リンクを押せば、認識した構造式を化合物データベース(PubChem)で検索した結果が表示されます。
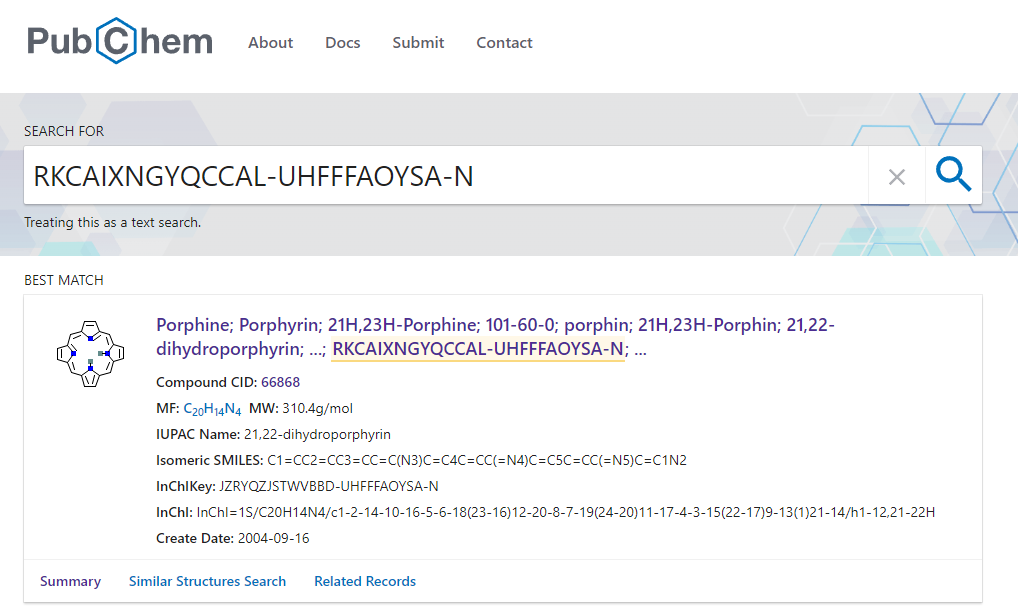
検索結果が表示されました。
データベースに登録されている化合物の場合、一番上に最も良く一致した化合物が表示されるのでリンクをクリックします。
化合物のページに移動しました。Porphyrin (ポルフィリン) です。
画像の構造式を正しく認識して正解の化合物に無事たどり着けました。
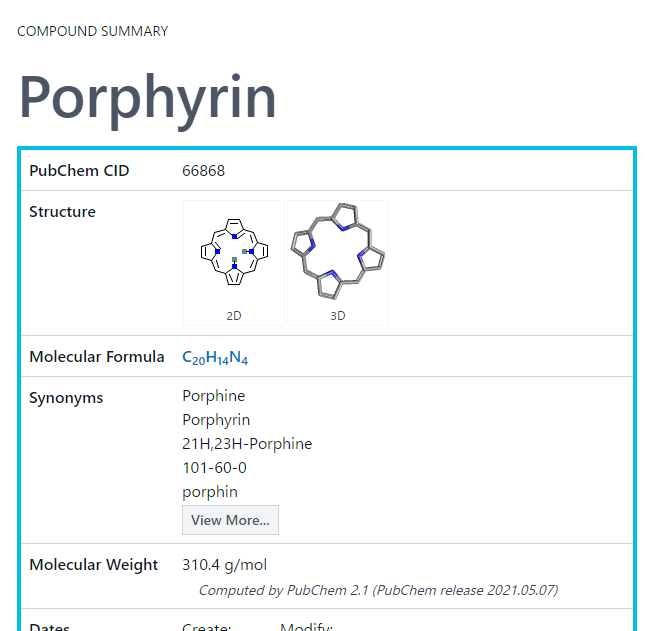
一致する化合物が見つからないときは
構造式の認識結果が誤っている場合も、PubChem側の類似化合物検索機能でお目当ての化合物を見つけられる場合があります。
例えば以下の認識結果の場合、一部の水素原子が誤って重水素 (²H) になってしまっているため、検索リンクを押しても化合物がヒットしません。
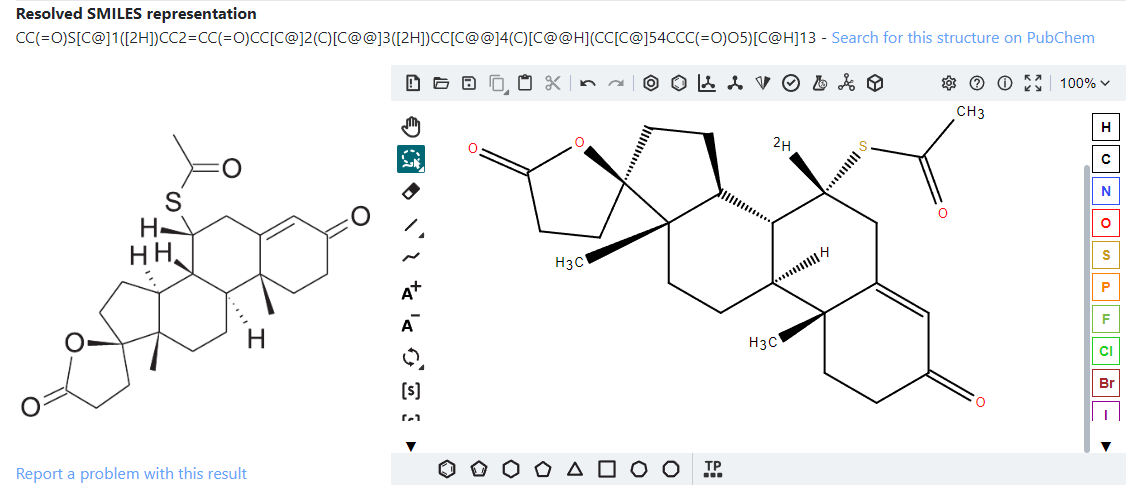
そのような場合は認識結果の SMILES文字列をコピーして検索ボックスに貼り付け、改めて検索し直します。
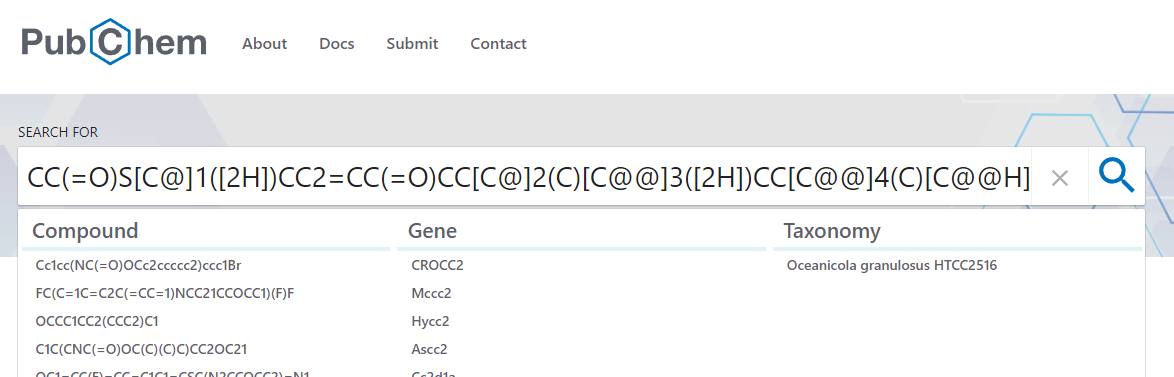
検索結果の Similarity タブをクリックすると、類似する構造の化合物が表示されます。
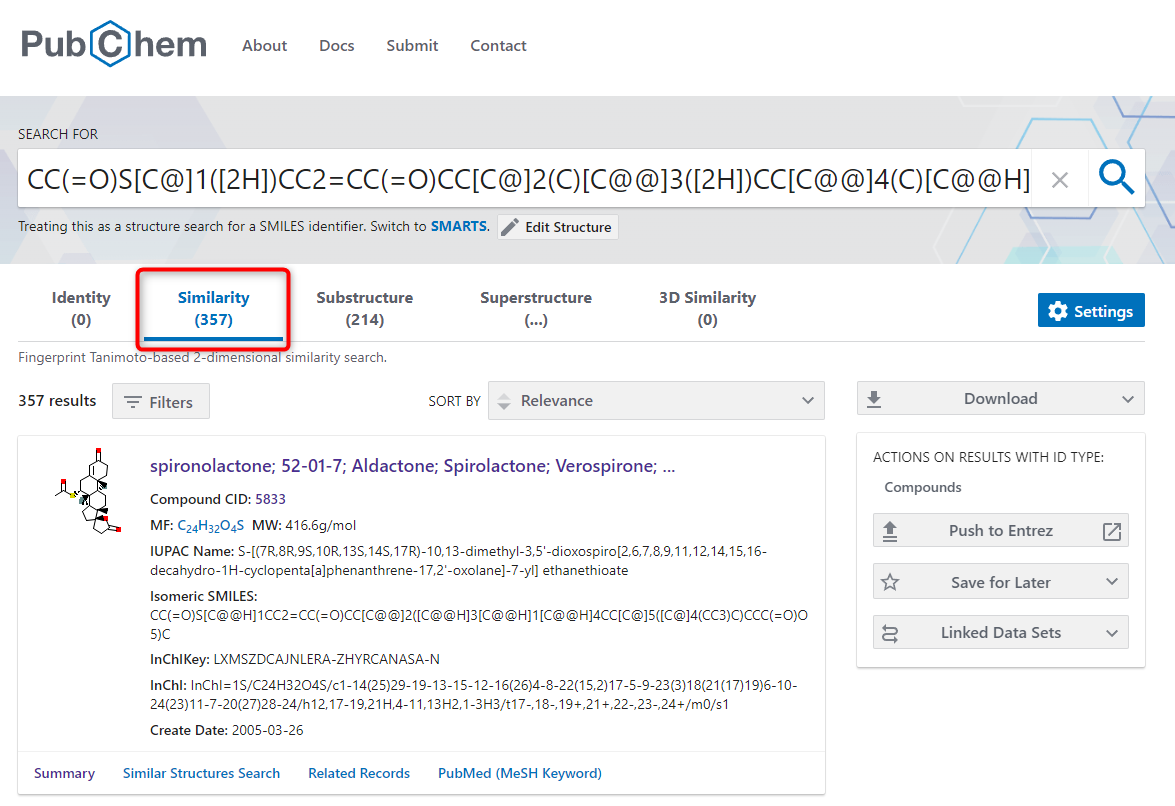
ヒットした化合物のページに移動し、Structures「構造」の項を見て元の構造式と一致しているか確認してみます。
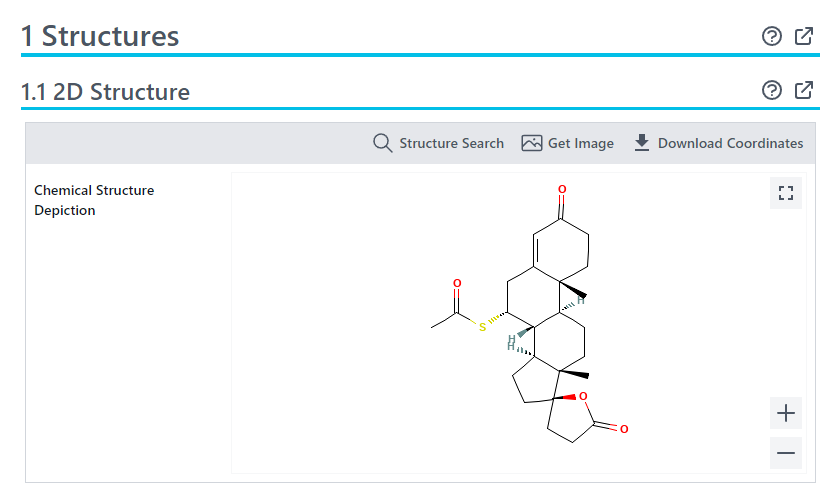
向きは異なりますが認識対象画像の構造式と同じ構造でした。これで画像の構造式は spironolactone (スピロノラクトン) と特定できました。
PDF中の構造式を一括認識
論文PDFの中に複数の構造式が含まれている場合、それらを一括で認識して一つひとつ表示させることもできます。
例えば以下のように文章の中に混在する複数の構造式であっても、自動で個々を認識できます。
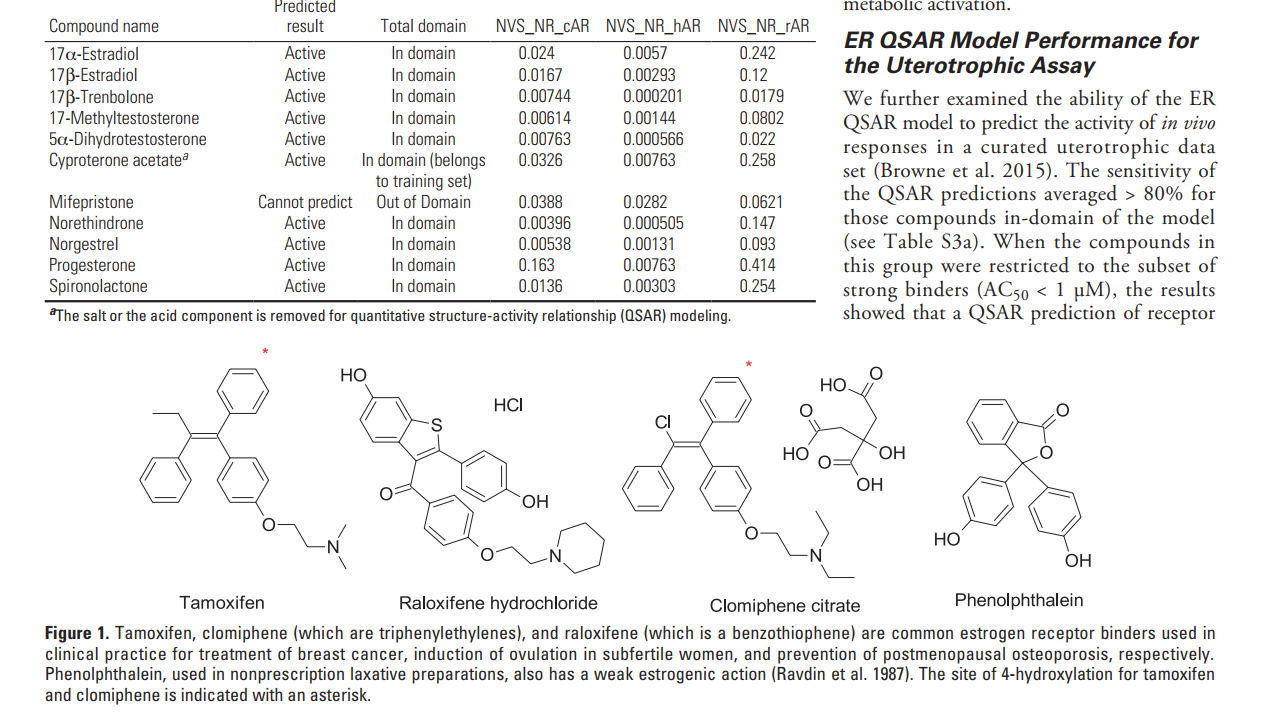
今回試しに利用したPDFはこちらの論文です。(パブリックドメイン)
まず、赤矢印で示したボタンのところに構造式を含むPDFファイルをドラッグ&ドロップするか、ボタンを押してファイル選択ダイアログを表示させてそこからPDFを選んでください。

すると認識処理が始まり、PDF中の構造式の認識結果が一覧で表示されます。
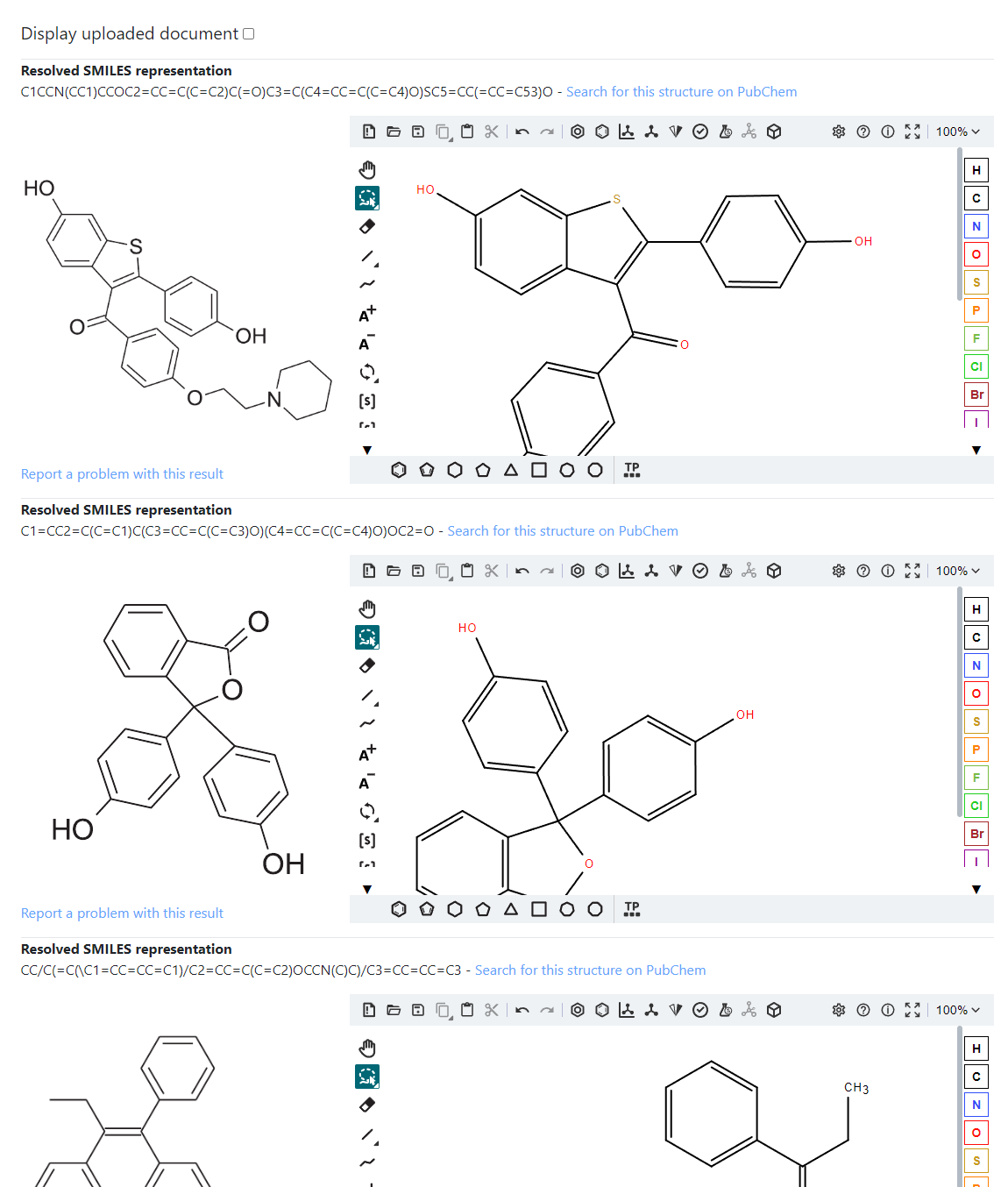
※構造式の順番はPDFでの登場順とは多少前後する場合があります。
※画像のように構造式エディタで構造式の一部分しか表示されていない場合は、エディタ右上のズーム倍率ボタンを押して Zoom out を押せば縮小して全体を表示できます。
構造式の認識精度は凄まじく、冒頭の4つの構造式を全て正しく認識できていました。
(Clomiphene citrate「クロミフェンクエン酸塩」はクロミフェンとクエン酸が別々に認識されていました)
ステロイド化合物の認識で微妙に間違っていましたが、先ほど説明したように認識結果の SMILES文字列をコピーしてPubChemで再検索して Similarity から正しい化合物を見つける方法で問題なく対応できました。
認識した構造式を IUPAC名 に変換
画像認識結果ページの一番上にある Generate IUPAC names を押すと、IUPAC名への変換処理が走ります。
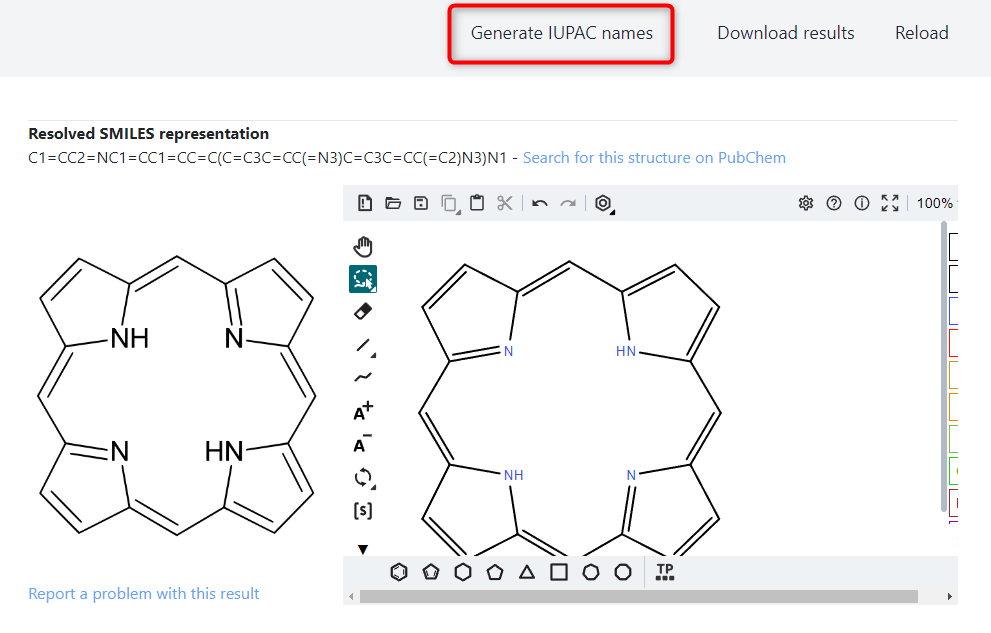
処理が終わると、SMILES記法から IUPAC名 への変換結果が表示されます。
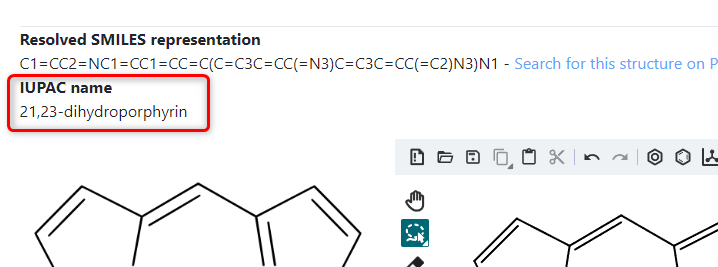
※ポルフィリンの場合はその骨格自体が母核になるので、IUPAC名もほぼそのままでした。
(21,23-dihydro というのは 21位/23位 の窒素原子に結合した水素原子を表しています)
試しに別の化合物で IUPAC名への変換を試してみました。(メバロン酸)
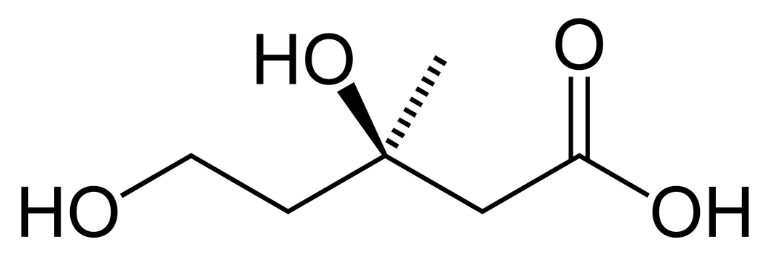
(3R)-3,5-dihydroxy-3-methylpentanoicacid「(3R)-3,5-ジヒドロキシ-3-メチルペンタン酸」と正しく命名できています。
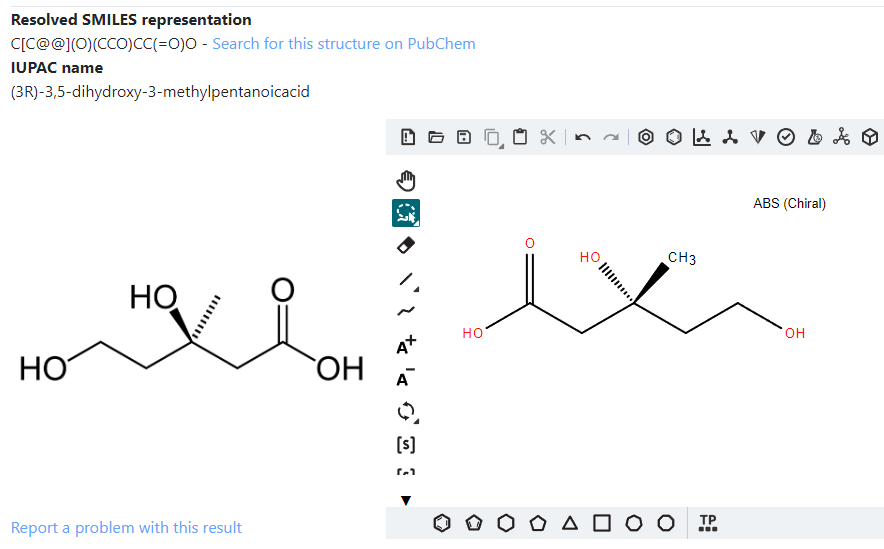
なおこのように構造式中に立体表記の破線-くさび形表記が含まれている場合、複雑な化合物ではそこそこの頻度で立体構造を逆に認識してしまうことがあるのでご注意ください。
(ペニシリン G を試したら一部逆に認識されました。また、不飽和脂肪酸のシス-トランスも逆になってしまいやすいです…。)
認識した構造式をその場でエディタ編集する
構造式エディタには画像認識で得られた構造式が表示されていますが、その場でこれを編集することができます。
認識結果が一部誤っている場合に自前で修正し、SMILES文字列を出力してPubChemで再検索すれば知りたかった物質にたどり着くことができます。
何かしらの構造式を自分で描きたい場合も、一から描く代わりに似た構造式を画像認識で読み込ませてエディタ編集するのが手っ取り早いです。
エディタの操作方法
表示されているエディタは Ketcher という Web ベースの化学構造エディターです。
使い方については以下のサイトの日本語チュートリアルが分かりやすいのでご参照ください。
Ketcherチュートリアル1 ドデシルベンゼンスルホン酸ナトリウムを描く
ㅤ
【他のチュートリアル】
・化学構造式の保存・画像形式での出力
・SMILESやInchlで構造を読みだす
・分子量や精密質量の計算
認識結果の構造式を修正して再検索する
例として以下のベンジルペニシリン (Penicillin G) の構造式画像を読み込ませ、誤認識している箇所を修正して再検索してみます。
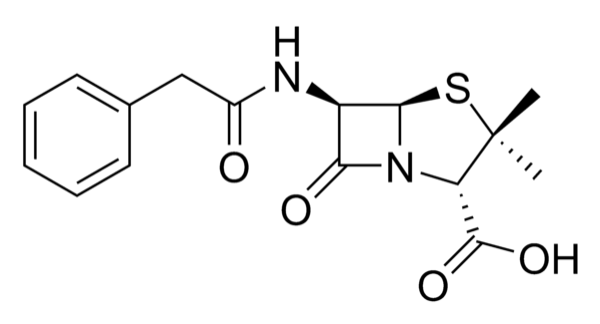
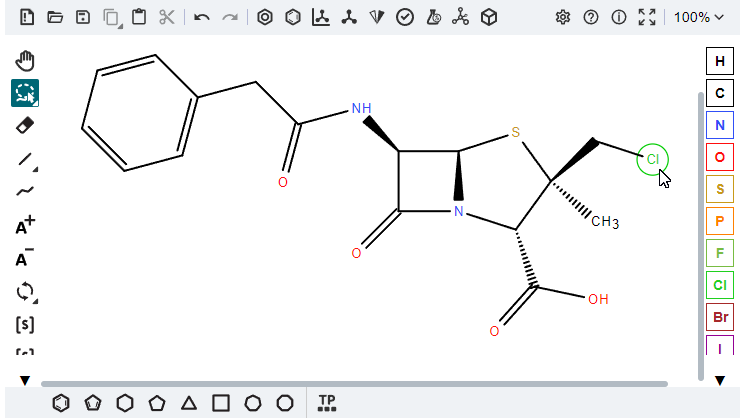
認識の結果、下記画像のマウスカーソル部分に余計な塩素原子が付与されていました。
この塩素原子をクリックして Delete キーを押し、塩素原子を消します。
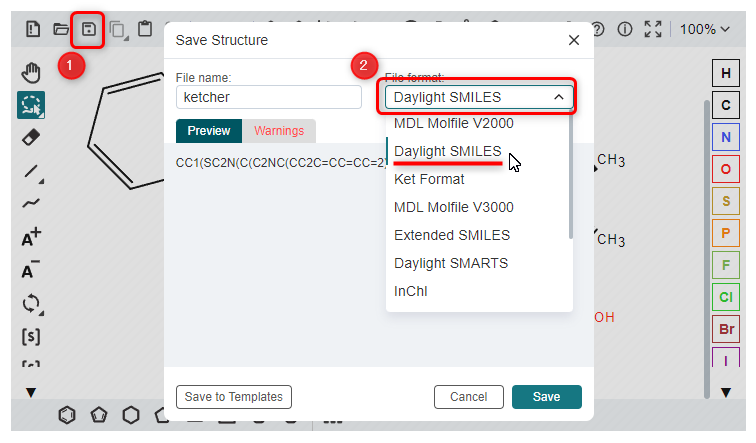
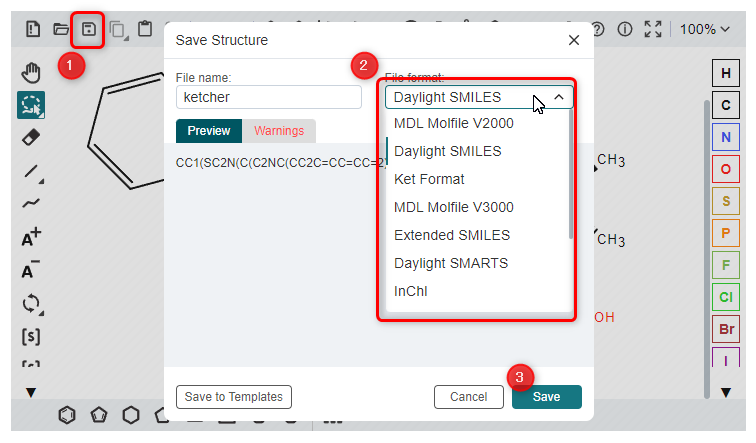
修正が完了したら以下の手順で構造式を SMILES文字列に変換します。
- 左上の保存ボタンを押す
- File format から Daylight SMILES を選ぶ
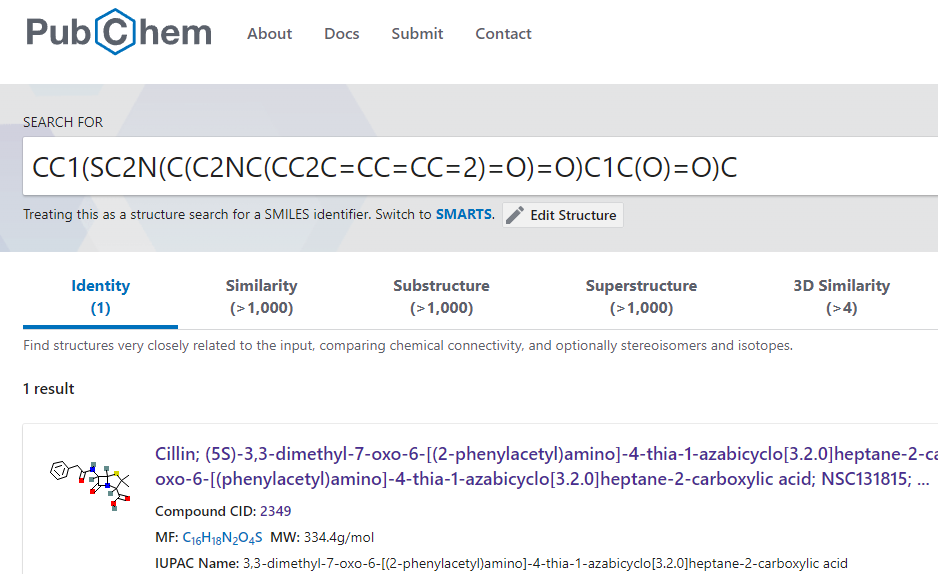
表示されたSMILES文字列をコピーして PubChem で検索すれば、お目当ての物質にたどり着けます。
認識した構造式を好きな形式で保存する
エディタの保存ボタンから好きな形式で構造式を保存できます。
- 画像として保存したい
→ PNG Image - ベクター画像
→ SVG Document - .mol ファイル
→ MDL Molfile V3000 - SMILES記法
→ Daylight SMILES
その他情報
DECIMER プロジェクトとは
DECIMER は The Deep Learning for Chemical Image Recognition「化学画像認識のためのディープラーニング」の略称であり、出版された化学文献から化学構造を検出・認識できるオープンソースで無料利用可能なツールを提供することで、オープンサイエンスを支援することを目的としたプロジェクトです。
DECIMER プロジェクト の概要は以下のリンク先ページで説明されています。
リンク:Deep Learning for chemical information mining
ツールの研究・開発を行っているのはドイツの フリードリヒ・シラー大学イェーナ (Friedrich-Schiller-University of Jena) の ケモインフォマティクス・計算機メタボロミクスの研究室です。
研究室の公式ページリンク:Cheminformatics and Computational Metabolomics
このような素晴らしいツールをオープンソースかつ誰でも無料で簡単に利用できる状態で提供してくださり、ありがたい限りです。
DECIMER の論文
このプロジェクトの技術開発にあたり出版されている論文のリストがこちらになります。
- DECIMER.ai: an open platform for automated optical chemical structure identification, segmentation and recognition in scientific publications
Rajan, K., Brinkhaus, H.O., Agea, I.A. Zielesny, A., Steinbeck, C. Nat Commun, 14, 5045 (2023). - DECIMER: towards deep learning for chemical image recognition
Rajan, K., Zielesny, A., Steinbeck, C. J Cheminform, 12, 65 (2020). - DECIMER-Segmentation: Automated extraction of chemical structure depictions from scientific literature
Rajan, K., Brinkhaus, H.O., Sorokina, M. et al. J Cheminform, 13, 20 (2021). - DECIMER 1.0: deep learning for chemical image recognition using transformers
Rajan, K., Zielesny, A., Steinbeck, C. J Cheminform, 13, 61 (2021). - STOUT: SMILES to IUPAC names using neural machine translation
Rajan, K., Zielesny, A., Steinbeck, C. J Cheminform, 13, 34 (2021).